pandas, matplotlib, seaborn 모듈 활용하기
06 Jan 2018 | 머신러닝 Python여기서 사용한 테스트 데이터는 여기에서 받을 수 있습니다.
Pandas
pandas는 많은 데이터를 쉽게 다룰 수 있는 기능을 제공하는 Python 모듈입니다.
예제 코드
import pandas as pd
data_set = pd.read_csv('pima-indians-diabetes.csv',
names=["pregnant", "plasma", "presure", "thickness",
"insulin", "BMI", "pedigree", "age", "class"])
print(data_set.head(5))
print('====================================================================\n')
print(data_set.info())
print('====================================================================\n')
print(data_set.describe())
data_set.head(5)는 상위 5개의 데이터들의 리스트를 조회하는 명령어입니다. 또한 info() 함수를 이용해서 각 정보들의 데이터 타입이나 데이터 개수 등을 확인할 수 있고, describe() 함수를 이용해서 정보별 샘플의 개수, 평균, 표준 편차, 최소/최대값 등을 조회할 수 있습니다.
실행 결과
pregnant plasma presure thickness insulin BMI pedigree age class
0 6 148 72 35 0 33.6 0.627 50 1
1 1 85 66 29 0 26.6 0.351 31 0
2 8 183 64 0 0 23.3 0.672 32 1
3 1 89 66 23 94 28.1 0.167 21 0
4 0 137 40 35 168 43.1 2.288 33 1
==================================================================================
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 768 entries, 0 to 767
Data columns (total 9 columns):
pregnant 768 non-null int64
plasma 768 non-null int64
presure 768 non-null int64
thickness 768 non-null int64
insulin 768 non-null int64
BMI 768 non-null float64
pedigree 768 non-null float64
age 768 non-null int64
class 768 non-null int64
dtypes: float64(2), int64(7)
memory usage: 54.1 KB
None
==================================================================================
pregnant plasma presure thickness insulin BMI \
count 768.000000 768.000000 768.000000 768.000000 768.000000 768.000000
mean 3.845052 120.894531 69.105469 20.536458 79.799479 31.992578
std 3.369578 31.972618 19.355807 15.952218 115.244002 7.884160
min 0.000000 0.000000 0.000000 0.000000 0.000000 0.000000
25% 1.000000 99.000000 62.000000 0.000000 0.000000 27.300000
50% 3.000000 117.000000 72.000000 23.000000 30.500000 32.000000
75% 6.000000 140.250000 80.000000 32.000000 127.250000 36.600000
max 17.000000 199.000000 122.000000 99.000000 846.000000 67.100000
pedigree age class
count 768.000000 768.000000 768.000000
mean 0.471876 33.240885 0.348958
std 0.331329 11.760232 0.476951
min 0.078000 21.000000 0.000000
25% 0.243750 24.000000 0.000000
50% 0.372500 29.000000 0.000000
75% 0.626250 41.000000 1.000000
max 2.420000 81.000000 1.000000
matplotlib, seaborn
matplotlib은 Python에서 그래프를 그리기 쉽도록 해주는 모듈입니다. 그리고 seaborn 모듈은 각 정보들끼리의 상관관계를 쉽게 볼 수 있도록 해줍니다.
Heatmap 예제 코드
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
data_set = pd.read_csv('pima-indians-diabetes.csv',
names=["pregnant", "plasma", "presure", "thickness",
"insulin", "BMI", "pedigree", "age", "class"])
# 그래프의 크기 지정
plt.figure(figsize=(8, 8))
# Heatmap 설정
sns.heatmap(data_set.corr(), linewidths=0.1, vmax=0.5, cmap=plt.cm.gist_heat,
linecolor='white', annot=True)
# 렌더링
plt.show()
실행 결과
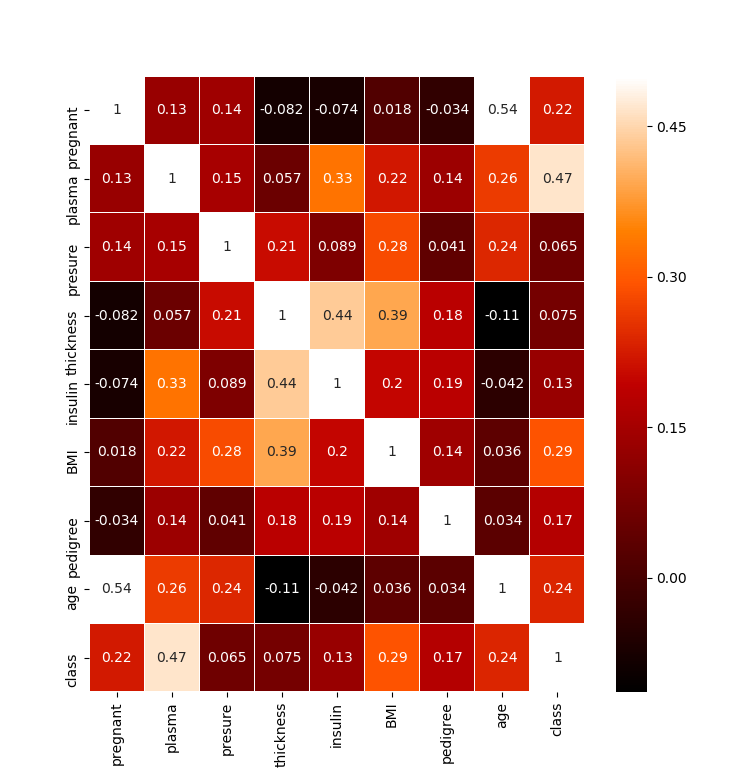
Heatmap을 이용하면 각 속성들간의 상관관계 크기를 알 수 있습니다. 위 결과에서는 plasma 항목과 class의 항목의 상관관계가 높은 것을 확인할 수 있습니다.
Grid 예제 코드
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
data_set = pd.read_csv('pima-indians-diabetes.csv',
names=["pregnant", "plasma", "presure", "thickness",
"insulin", "BMI", "pedigree", "age", "class"])
grid = sns.FacetGrid(data_set, col='class')
grid.map(plt.hist, 'plasma', bins=10)
plt.show()
실행 결과
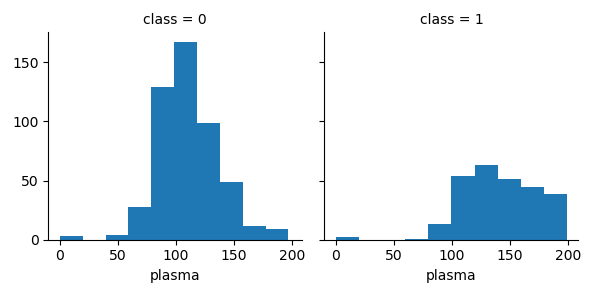
위 그래프를 보면 class 속성이 1인 경우(위 데이터는 당뇨병 환자를 의미하는 속성) plasma 항목의 수치가 150 이상일 때, class = 0일 때의 경우와 다르다는 것을 확인할 수 있습니다.
